1月2日,《Nature Communications》在线发表了我院沈其荣院士团队LorMe实验室与西南大学张勇教授(资源环境学院/长江经济带绿色农业发展研究中心)合作的最新研究成果《Effects of plant tissue permeability on invasion and population bottlenecks of a phytopathogen》。该研究通过建立等位基因标记技术,动态示踪微生物种群变化,突破了根际入侵过程中无法精细刻画致病菌种群演替的关键技术瓶颈,解析了根际免疫制约土传致病菌入侵的过程与微生态机制,为土传致病菌的生态阻控提供理论和技术支撑。
致病菌是制约土壤健康和粮食安全的典型生物污染因子。生产上,常采用土壤熏蒸、农药抗生素、抗性品种来阻控土传致病菌。尽管当季可取得一定效果,但致病菌遗传多样性丰富、适应能力强,极易产生适应性进化,导致抗生素失效、作物抗性退化。当前,致病菌的多样性和生态进化研究已经成为土壤生物健康领域的前沿热点。然而,研究这一问题的难点在于如何动态追踪致病菌的种群演替规律。有研究尝试利用荧光基因和抗性标记等分子手段来评估致病菌的种群数量,这增加了被标记细胞的代谢负担,更存在稳定性差、通量低等问题,难以实现复杂土壤-植物系统环境下致病菌动态的原位追踪。因此,研发高通量精准追踪技术,对精细刻画致病菌种群动态演替至关重要,有利于制定和优化土传致病菌的生态防控策略,对土壤生物健康培育具有重要意义。
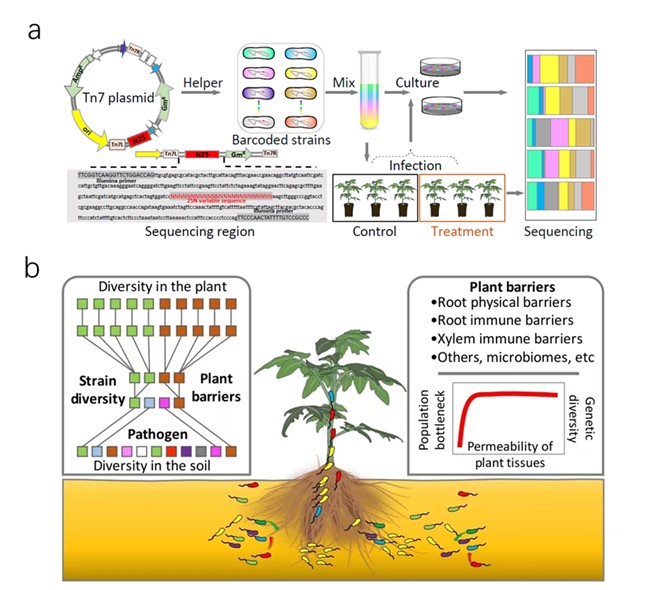
根-土交界面——根际抵御土传致病菌进入植物体内的天然屏障。根际微生物、根层细胞、免疫蛋白、拮抗物质等共同决定根际抵御土传病原菌入侵的能力,即根际免疫。然而,当前仍缺乏对根际免疫强度的有效评估方法,更缺乏对根际免疫制约致病菌入侵过程的深刻理解。为此,LorMe实验室以土传植物病原细菌青枯菌为研究对象,联合西南大学资源环境学院张勇教授课题组协同创新,建立了高通量基因组等位基因标记技术:在青枯菌染色体的非编码区插入序列随机的DNA片段,人工构建了等位基因标记的青枯菌复合群体,通过扩增子测序检测DNA片段的丰度变化,采样群体遗传学算法量化根际免疫,利用生态模型探究青枯菌的种群动态变化。为评估根际免疫强度对致病菌入侵过程的影响,本研究以抗性和易感两种番茄品种为材料,采用灌根接种、伤根接种和注射接种三种方式,模拟根际免疫强度的梯度变化,探究根际入侵前后,标记青枯菌种群结构、组成和多样性的变化规律。研究发现根际免疫强度的增加,选择压增强,导致成功入侵植物体内青枯菌的种类和多样性减少。其中,根系物理结构和免疫能力的完整性,阻控青枯菌入侵的关键。该研究揭示了根际免疫制约土传致病菌入侵过程与微生态机制,为土传致病菌的生态阻控提供理论与技术支撑。
该研究得到国家自然科学基金项目(重大项目、杰出青年项目、面上项目等)、中央高校基本业务费和江苏省优秀青年基金等项目的资助。我院江高飞副教授为第一作者,韦中教授和西南大学张勇教授为共同通讯作者。我院沈其荣院士、赵方杰教授和徐阳春教授,西南大学陈新平教授、石孝均教授、丁伟教授,宾夕法尼亚大学Francisco Dini-Andreote教授等参与指导了该项研究。
全文链接:https://doi.org/10.1038/s41467-023-44234-7

 微信公众号
微信公众号